Statystyczna Eksploracja Danych
LABORATORIUM 9
Z uczeniem bez nadzoru mamy do czynienia wtedy, gdy oryginalne dane nie posiadają przypisania do konkretnych klas tak, jak jest to np. w przypadku drzew decyzyjnych. Nie następuje tu więc proces uczenia się klasyfikatora na podstawie konkretnych przypadków.
Jednym z szerokich tematów w uczeniu bez nadzoru jest grupowanie, również nazywane analizą skupień czy klasteryzacja (od ang. cluster analysis). Podstawową ideą tego podejścia jest zbieranie (grupowanie) elementów tak, aby utworzyć (dość) jednorodne klasy. W ramach dzisiejszych zajęć zajmiemy się dwiema metodami: k-średnich oraz hierarchiczną.
>
METODA K-ŚREDNICH
Metoda k-średnich (ang. k-means), została szerzej omówiona na Wykładzie 7. Jej założenia są następujące:
- mamy \(n\)-elementowy zbiór obserwacji \(\mathbf{x}_i\) przestrzeni \(\mathbb{R}^p\),
- chcemy podzielić tę próbę na \(K\) skupień,
- rozpatrujemy sumę kwadratów odegłości \(d_{ij} = d(\mathbf{x}_i, \mathbf{x}_j)\) pomiędzy parami punktów \(T = \frac{1}{2}\sum\limits_{i=1}^n\sum\limits_{j=1}^nd_{ij}\),
- sumę \(T\) mozna rodzielić na \(T = W + B\), gdzie \(W = \frac{1}{2}\sum\limits_{k=1}^K\sum\limits_{C(i)=k}\sum\limits_{C(j)=k}d_{ij}\), a \(B = \frac{1}{2}\sum\limits_{k=1}^K\sum\limits_{C(i)=k}\sum\limits_{C(j) \neq k}d_{ij}\), \(W\) jest odległością wewnątrz skupień, a \(B\) - pomiędzy nimi,
- zadaniem jest minimalizacja \(W\) (lub maksymalizacja \(B\)).
W praktyce algorytm metody k-średnich inicjalizuje środki \(K\) skupień (gdzie \(K\) jest narzucone z góry) i w kolejnych iteracjach przypisuje najbliższe punkty do tych skupień oraz wyznacza nowe skupienia itd. aż w układzie nie będzie zachodzić żadna zmiana.
Do wizualizacji metody k-średnich w pakiecie użyjemy funkcji kmeans() z biblioteki cluster:
# Generowanie
draw.data.gauss <- function(S1, S2, m1, m2, n1, n2) {
X1 <- mvrnorm(n1, m1, S1)
X2 <- mvrnorm(n2, m2, S2)
X1 <- data.frame(X1); colnames(X1) <- c("x", "y")
X2 <- data.frame(X2); colnames(X2) <- c("x", "y")
X1$class <- 1; X2$class <- 2
data <- rbind(X1, X2); data$class <- factor(data$class)
return(data)
}
# Rysowanie punktów
plot.data <- function(data) {
cols <- c("blue", "orange")
plot(data[,1:2], col = cols[data$class], cex = 3, pch = 19)
text(data[,1:2], labels = 1:nrow(data), cex = 1, col = "white", font = 2)
}
# Parametry danych z rozkładu Gaussa
S1 <- matrix(c(2, 0, 0, 2), 2, 2)
S2 <- matrix(c(2, 0, 0, 2), 2, 2)
m1 <- c(-1, -1)
m2 <- c(1, 1)
n1 <- 10
n2 <- 10
# Ustawienie ziarna dla losowania danych
set.seed(1290)
# Generowanie obserwacji
data <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
# Definiowanie kolorów
blueop <- "#0000ff55"; orangeop <- "#ffa50055"; redop <- "#00ff0055"
# Algorytm k-średnich dla K=2
par(mfrow = c(1,2))
plot.data(data)
title("Oryginalne dane")
km2 <- kmeans(data[,1:2], 2)
plot.data(data)
title("Oryginalne dane + efekty k-średnich")
points(data[,1:2], pch = 19, cex = 4, col = c(blueop, orangeop)[km2$cluster])
Rysunek 8.1
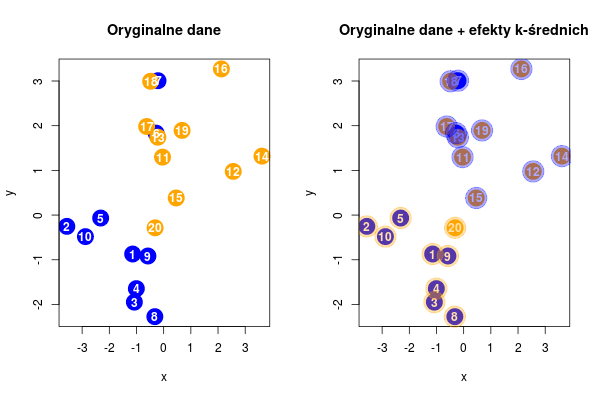
Oczywiście liczba \(K=2\) została przez nas z góry narzucona. Równie dobrze można zażądać wyszukiwania 3 skupień. Przy okazji możemy też zwizualizować fakt, że algorytm nie zawsze prowadzi do tych samych wyników.
# Algorytm k-średnich dla K=3
plot.clust <- function(n, data, K) {
km <- kmeans(data[,1:2], K)
plot(data[,1:2], pch = 19, cex = 3, col = c("blue", "orange", "green")[km$cluster])
text(data[,1:2], labels = 1:nrow(data), cex = 1, col = "white", font = 2)
title(n)
return(km$tot.withinss)
}
par(mfrow = c(3,3))
W <- sapply(1:9, function(n) plot.clust(n, data, 3))
Rysunek 8.2
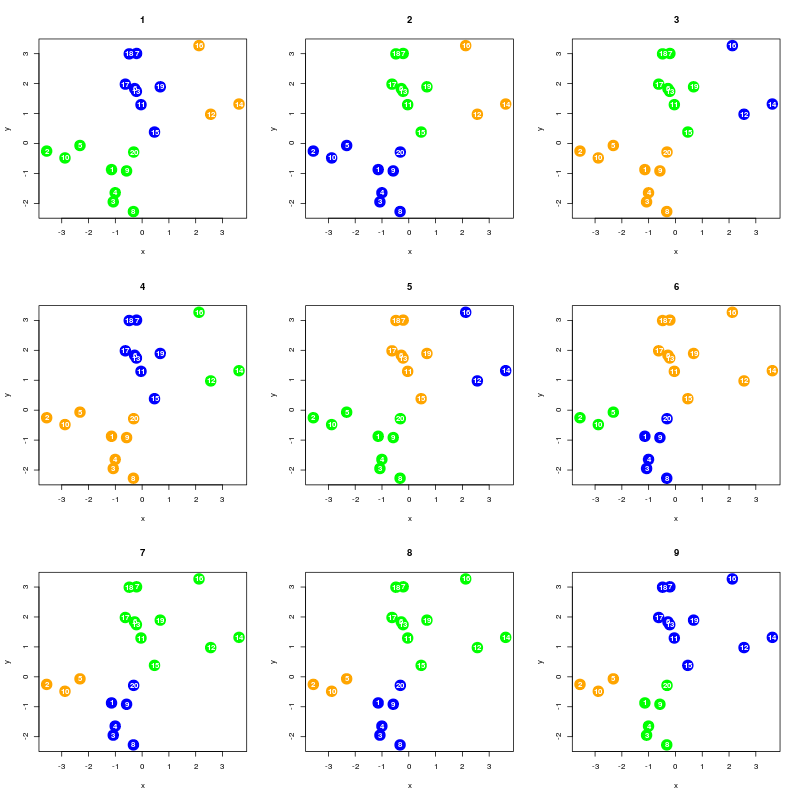
Jak widać, w niektórych przypadkach mamy do czynienia jedynie ze zmianą numeracji klasy (czyli kolorów). Jednak są przypadki, dla których podział na klastry daje znacząco inne efekty. Aby wybrać optymalny podział korzystamy z pola $tot.withinss oddającego wartość \(W\).
print(W)
26.92515 26.92515 26.92515 26.92515 26.92515 32.98386 32.98386 32.98386 32.98386
W przedstawionym przypadku realizacje 1-5 są optymalne (prawdopodobnie).
OPTYMALNA LICZBA SKUPIEŃ
Patrząc na wyniki z poprzedniej części powstaje dość oczywiste pytanie: jaka wartość \(K = K^*\) jest optymalna dla danego zbioru? Do wyznaczenia \(K^{*}\) wykorzystamy tzw. statystykę odstępu (ang. gap statistics). Poniżej dane, z których będziemy korzystać - bardzo podobne do tych, z których korzystaliśmy poprzednio.
library(Hmisc)
# Macierz kowariancji
S <- matrix(c(1,0,0,1),2,2)
# Wartosci średnie dla poszczególnych skupień
m1 <- c(4, 4)
m2 <- c(1, 1)
m3 <- c(4,-1)
kolory <- c(rep("orange", 30), rep("blue", 30), rep("green", 30))
# Losowanie punktów do klastrów
clust1 <- mvrnorm(30, m1, S)
clust2 <- mvrnorm(30, m2, S)
clust3 <- mvrnorm(30, m3, S)
# Zebranie punktów razem
all_points <- rbind(clust1, clust2, clust3)
# Wyznaczenie wartości min i max dla współrzędnych
xrange <- range(all_points[,1])
yrange <- range(all_points[,2])
xmin = xrange[1]; xmax = xrange[2]
ymin = yrange[1]; ymax = yrange[2]
# Rysowanie danych
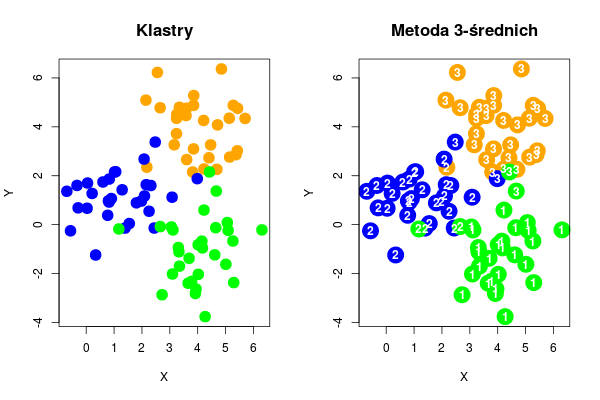
par(mfrow = c(1,2))
plot(all_points, col=kolory, pch=19, cex=2, xlab="X", ylab="Y", xlim=c(xmin,xmax), ylim=c(ymin,ymax))
title("Klastry", cex.main=1.4, font=2)
# Wykonanie metody k-średnich dla k=3
cl <- kmeans(all_points, 3)
# Nadrukowanie na klastrach przynależnosci do klas
# otrzymanej z metody k-średnich
plot(all_points, col=kolory, pch=19, cex = 3, xlab="X", ylab="Y", xlim=c(xmin,xmax), ylim=c(ymin,ymax))
text(all_points[,1], all_points[,2], cl$cluster, col = "white", font=2)
title("Metoda 3-średnich", cex.main=1.4, font = 2)
Rysunek 8.3
Punktem odniesienia w metodzie statystyki odstępu są dane losowe: w pudełku o wymiarach wyznaczonych przez skrajne współrzędne losujemy taką samą liczbę punktów co w danych oryginalnych lecz korzystamy z rozkładu jednorodnego.
# Dane losowe
rnd_points <- all_points
# Losowanie danych z rozkladu jednostajnego
rnd_points[,1] <- runif(90, xmin, xmax)
rnd_points[,2] <- runif(90, ymin, ymax)
# Rysowanie danych losowych
plot(rnd_points, pch=19, cex = 2, xlim=c(xmin,xmax), ylim=c(ymin,ymax), xlab="X", ylab="Y")
title("Dane losowe", cex.main=1.4, font=2)
Rysunek 8.4
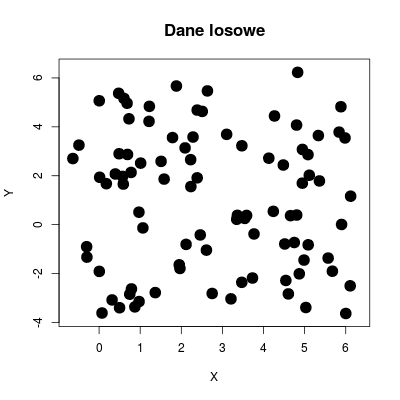
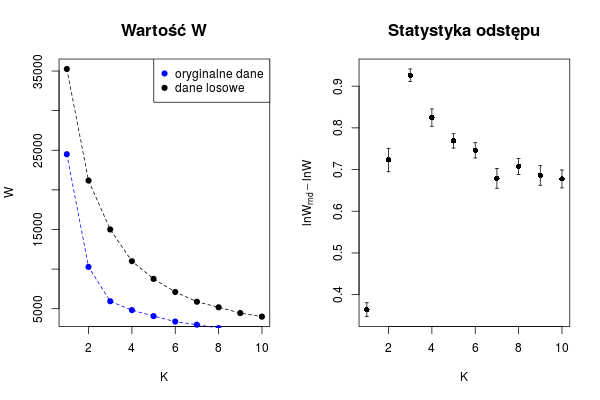
Wreszcie przechodzimy do kluczowego etapu procedury. Jak widać poniżej sama wartość \(W\) jest niewystarczająca, gdyż po prostu spada wraz ze wzrostem \(K\). W efekcie z dla każdej wartości \(K\) liczmy \(W\) z danych oryginalnych, a następnie tworzymy robimy to samo dla danych losowych (przy czym powtarzamy to \(N\) razy) wyznaczając \(\langle W_{rnd} \rangle = \sum_{i=1}^N W^i_{rnd}\). Staystyka odstępu dla danego \(K\) jest zdefiniowana jako \(\ln \langle W_{rnd} \rangle - \ln W\), a optymalną wartość \(K = K^*\) bierze się z maksimum wykresu.
# Obliczanie statystyki odstepu
# Mamy od 1 do K skupien
# Dla kazdego skupienia przeprowadzamy N prob
K = 10
N = 20
# Deklaracja macierzy, w ktorych trzymamy wyniki
# z kolejnych interacji
dane <- array(0, dim = c(K, N))
dane_rnd <- array(0, dim = c(K, N))
# Główna pętla
for(c in 1:K)
{
for(n in 1:N)
{
cl <- kmeans(dist(all_points), c)
rnd_points[,1] <- runif(90, xrange[1], xrange[2])
rnd_points[,2] <- runif(90, yrange[1], yrange[2])
cl_rnd <- kmeans(dist(rnd_points), c)
dane[c,n] <- cl$tot.withinss
dane_rnd[c,n] <- cl_rnd$tot.withinss
}
}
# Wyznaczenie wartości średnich dla danych i danych losowych
# oraz odchyleń dla danych losowych
avgW <- apply(dane, 1, mean)
avgWrnd <- apply(dane_rnd, 1, mean)
sdWrnd <- apply(dane_rnd, 1, sd)
# Rysowanie statystki odstępu
par(mfrow = c(1,2))
plot(1:K, avgWrnd, pch = 19, t="o", lty = 2, ylab = "W", xlab = "K")
points(1:K, avgW, pch = 19, t="o", lty = 2, col = "blue")
title("Wartość W", cex.main=1.4, font=2)
legend("topright", c("oryginalne dane", "dane losowe"), col = c("blue", "black"), pch = 19)
errbar(1:K, log(avgWrnd/avgW), log(avgWrnd+sdWrnd/sqrt(20))-log(avgW), log(avgWrnd-sdWrnd/sqrt(20))-log(avgW), xlab="K", ylab=expression(lnW[rnd]-lnW))
title("Statystyka odstępu", cex.main=1.4, font=2)
Rysunek 8.5
METODY HIERARCHICZNE
Innym posobem wykonania analizy skupień jest wykorzystanie metod hierarchicznych. Ich ogólny opis można zawrzeć w następujących punktach:
- opierają sie na pomiarze uogólnionej odmienności między dwoma dowolnymi zbiorami obserwacji,
- nie wymagają z góry określenia liczby skupień,
- w pierwszym kroku metody algomeracyjnej tworzymy tyle skupień, ile jest obserwacji,
- w nastepnym kroku w jedno skupienie łączona jest para najmniej odległych obserwacji,
- z koroku na krok skupień jest coraz mniej, aż w ostatnim powstaje cała próba w jednym skupieniu,
- w efekcie otrzymuje się nieskierowane drzewo (dendrogram).
Poniżej pokazany został prosty dendrogram dla danych z początku zajęć.
# Parametry danych z rozkładu Gaussa
S1 <- matrix(c(2, 0, 0, 2), 2, 2)
S2 <- matrix(c(2, 0, 0, 2), 2, 2)
m1 <- c(-1, -1)
m2 <- c(1, 1)
n1 <- 10
n2 <- 10
# Ustawienie ziarna dla losowania danych
set.seed(1290)
# Generowanie obserwacji
data <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
# Rysowanie danych
par(mfrow = c(1,2))
plot.data(data); title("Dane")
# Tworzenie dendrogramu
hc <- hclust(dist(data[,1:2]))
# Rysowanie dendrogramu
plot(hc, hang = -1, ylab = "Odległość", xlab = "Numer obserwacji", main = "Dendrogram", sub = NA, cex = 0.9, font = 2)
# Obwódka klastrów
rect.hclust(hc, 2, border = c("blue", "orange"))
Rysunek 8.6
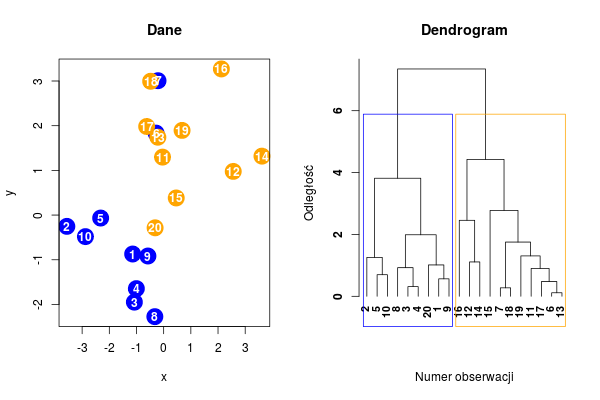
Kluczowe informacje dotyczące odległości, dla których tworzone są klastry oraz łączących się punktów są podane w polach $height oraz $merge (ujemne wartości oznaczają łączone obserwacje, natomiast dodatnie oddają klastry połączone w podanym kroku algorytmu - np. -17 1, to połączenie obserwacji nr 17 z klastrem powstałym w pierwszym kroku).
# Wektor połączeń
print(hc
$height)
[1] 0.1134074 0.2723499 0.3113741 0.4765396 0.5633944 0.6974915 0.9001276 0.9251001 1.0134697 1.1104037 1.2576203 1.3054874
[13] 1.7526470 1.9880677 2.4569027 2.7758910 3.8164530 4.4257007 7.3490213
# Dodanie linii na wysokości pierwszego połączenia
abline(
h = hc
$height[
1],
col = "red",
lty = 2)
# Połączenia
print(hc
$merge)
[,1] [,2]
[1,] -6 -13
[2,] -7 -18
[3,] -3 -4
[4,] -17 1
[5,] -1 -9
[6,] -5 -10
[7,] -11 4
[8,] -8 3
[9,] -20 5
[10,] -12 -14
[11,] -2 6
[12,] -19 7
[13,] 2 12
[14,] 8 9
[15,] -16 10
[16,] -15 13
[17,] 11 14
[18,] 15 16
[19,] 17 18
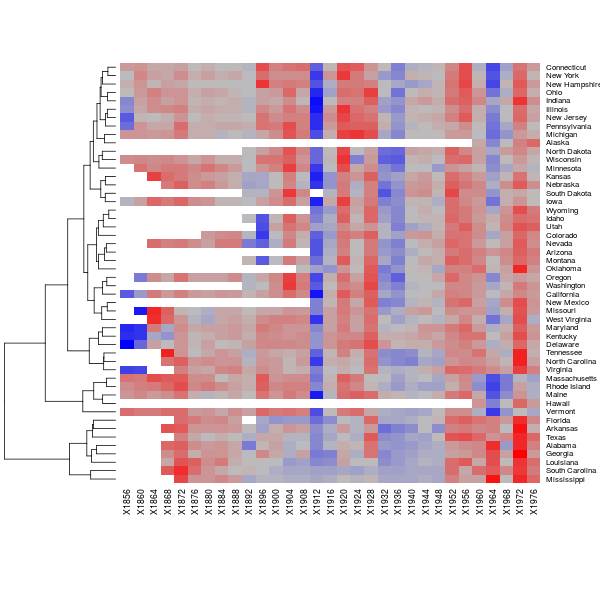
Bardzo spektakularnym sposobem wizualizacji danych jest funkcja heatmap. Umożliwia ona nie tylko prezentację samych trójwymiarowych danych ale także automatyczne stworzenie dendrogramów (opcja Colv=NA zapobiega tworzeniu dendrogramu dla kolumn). Jakbo zbiór danych posłuży nam zestawienie głosów oddanych na kandydatów Republikanów w wyobrach prezydenckich USA dla różnych lat i różnych stanów.
# Tworzenie gradientu kolorów
colfunc <- colorRampPalette(c("blue", "gray", "red"))
# Pobranie danych
votes.repub <- cluster::votes.repub
# Tworzenie wykresu
heatmap(as.matrix(votes.repub), col = colfunc(60), Colv = NA)
Rysunek 8.7