Statystyczna Eksploracja Danych
LABORATORIUM 7
W wielu przypadakch skuteczność rozpatrywanych klasyfikatorów nie jest dla nas satsfakcjonująca lub też okazuje się, że są one bardzo podatne na drobne zmiany. Szczególnie dotyczy to drzew. Jedną z metodą, która pozwala omijać te mankamenty jest tworzenie zespołów klasyfikatorów. Ogólna idea jest związana z prostym faktem: jeśli będziemy dysponowali zestawem \(M\) słabych klasyfikatorów (np. o ułamku błędnych klasyfikacji \(err = 0.45\)), to dla dużej wartości \(M\) decyzja podjęta przez większość będzie prawie na pewno prawdziwa. Oczywiście, tego typu twierdzenie dotyczt jedynie zespołu nieskorelowanych klasyfikatorów. Na dziesiejszych zajęciach poznamy trzy najpoularniejsze metody: bagging, boosting oraz lasy losowe.
BAGGING
Metoda agregacji bootstrapowej, czyli bagging, została szerzej omówiona naWykładzie 5. W tym miejscu przypomnimy jedynie sam algorytm:
- dla każdego \(m = 1,...M\) wylosuj pseudopróbę \(P_m\) z oryginalnej \(N\)-elementowej próby uczącej,
- na każdej pseudopróbie \(P_m\) naucz klasyfikator (np. drzewo) \(T_m\).
W efekcie posiadamy rodzinę \(M\) klasyfikatorów, każdy wyuczone na oddzielnej \(N\)-elementowej próbie powstałej poprzez metodę repróbkowania (bootstrap). Aby zaklasyfikować nową obserwację sprawdzamy jaka jest odpowiedź każdego z klasyfikatorów i wybieramy taką klasę, która została przypisana przez większość.
Po umieszczeniu bibliotek oraz standardowej funkcji związanej z rozkładem Gaussa a także rysowaniem
library(MASS)
library(rpart)
library(rpart.plot)
library(Hmisc)
# Generowanie
draw.data.gauss <- function(S1, S2, m1, m2, n1, n2) {
X1 <- mvrnorm(n1, m1, S1)
X2 <- mvrnorm(n2, m2, S2)
X1 <- data.frame(X1); colnames(X1) <- c("x", "y")
X2 <- data.frame(X2); colnames(X2) <- c("x", "y")
X1$class <- 1; X2$class <- 2
data <- rbind(X1, X2); data$class <- factor(data$class)
return(data)
}
# Rysowanie punktów
plot.data <- function(data) {
cols <- c("blue", "orange")
plot(data[,1:2], col = cols[data$class], cex = 2)
text(data[,1:2], labels = 1:nrow(data), cex = 0.6)
}
wybieramy parametry i tworzymy interesujące nas dane i wizualizujemy je
# Parametry danych z rozkładu Gaussa
S1 <- matrix(c(2, 0, 0, 2), 2, 2)
S2 <- matrix(c(2, 0, 0, 2), 2, 2)
m1 <- c(-0.25, -0.25)
m2 <- c(0.25, 0.25)
n1 <- 50
n2 <- 50
# Ustawienie ziarna dla losowania danych
set.seed(1290)
# Generowanie obserwacji
data <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
newdata <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
# Rysowanie danych
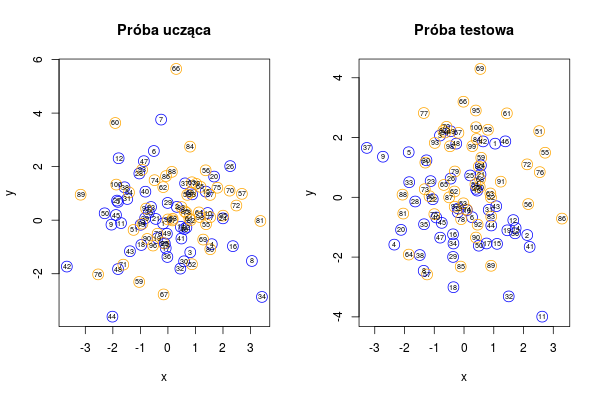
par(mfrow = c(1,2))
plot.data(data); title("Próba ucząca")
plot.data(newdata); title("Próba testowa")
Rysunek 7.1
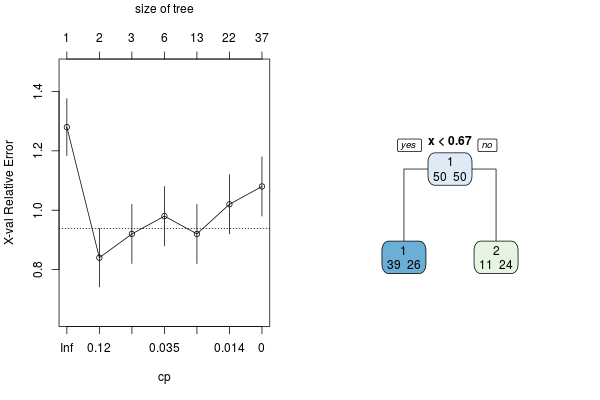
Ponieważ wartości oczekiwane klas są bardzo blisko siebie, a także losujemy po 50 elementów z każdej z klas, klasyfikator typu przycięte drzewo będzie miał dość niską skuteczność. Sprawdźmy to poniżej, wprowadzając funkcję obliczania błędu oraz przycinania drzew. Utworzymy też pełne drzewo, a następnie przytniemy je za pomocą algorytmu kosztu-złożoności (patrz poprzednie zajęcia). Poza tym wyznaczone zostaną błędy klasyfikacji dla pełnego drzewa dla PT (\(err_0\)) oraz przyciętego dla PU (\(err_1\)) i PT (\(err_2\)).
# Wyznaczanie optymalnej wartości CP zgodnie z regułą 1-SE
choose.cp <- function(tree) {
n <- which.min(tree$cptable[, 4])
n.min <- min(which(tree$cptable[, 4] < tree$cptable[n, 4] + tree$cptable[n, 5]))
return(tree$cptable[n.min, 1])
}
# Wyznaczanie blędu klasyfikatora
err.rate <- function(org.class, pred.class) {
CM <- table(org.class, pred.class)
return(1 - sum(diag(CM)) / sum(CM))
}
# Tworzenie pełnego drzewa
tree0 <- rpart(class ~ ., data, minsplit = 0, minbucket = 0, cp = 0)
# Przycinanie drzewa
tree <- prune(tree0, cp = choose.cp(tree0))
err0 <- err.rate(newdata$class, predict(tree0, newdata, type = "class"))
err1 <- err.rate(data$class, predict(tree, data, type = "class"))
err2 <- err.rate(newdata$class, predict(tree, newdata, type = "class"))
Rysunek 7.2
W tym momencie zaimplementujemy wreszcie same funkcje do metody bagging: bagging.own() oraz bagging.own.pred(). Pierwsza z nich służy do trenowania klasyfikatora, druga - do doknywania przewidywania (de facto pierwsza z nich korzysta od razu również z drugiej do wyznaczenia błędu klasyfikacji na zbiorze treningowym).
# Funkcja do przeprowadzanie procedury bagging
bagging.own <- function(data, N) {
# Dane: losowanie z oryginalnej próby
dane <- replicate(N, sample(1:nrow(data), rep = T))
# tworzenie drzew
trees <- lapply(1:N, function(i) rpart(class ~ ., data = data[dane[,i],], maxdepth = 1))
tmp <- list(dane = dane)
tmp$N <- N
tmp$data <- data
tmp$trees <- trees
tmp1 <- bagging.own.pred(tmp, data)
tmp$trees.class <- tmp1$trees.class
tmp$votes <- tmp1$votes
tmp$class <- tmp1$class
tmp$err <- tmp1$err
return(tmp)
}
# Funkcja do przeprowadzania przewidywania za pomoca baggingu
bagging.own.pred <- function(bag, data) {
tmp <- list()
trees.class <- sapply(1:bag$N, function(i) predict(bag$trees[[i]], newdata = data, type = "class"))
votes <- t(sapply(1:nrow(trees.class), function(i) table(factor(trees.class[i,], levels = levels(data$class)))))
class <- factor(levels(data$class)[apply(votes, 1, which.max)], levels = levels(data$class))
tmp$trees.class <- trees.class
tmp$votes <- votes
tmp$class <- class
tmp$err <- err.rate(data$class, tmp$class)
return(tmp)
}
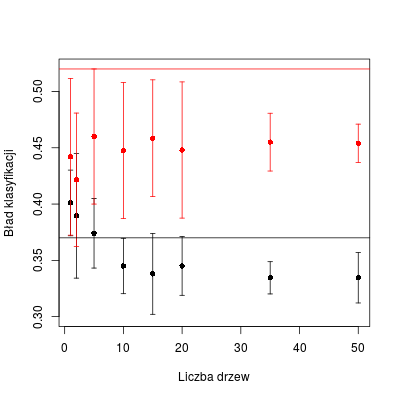
Finalnie wykonujemy po 10 realizacji dla każdego rozmiaru (tj. ilości klasyfikatorów w zespole) zarówno dla PU jak i PT.
# Liczba drzew
vals <- c(1, 5, 10, 20, 50)
# Wywołanie algorytmu bagging dla różnej liczby drzew dla PU (10 realizacji)
tab <- sapply(vals, function(v) replicate(10, bagging.own(data, v)$err))
# Wywołanie algorytmu bagging dla różnej liczby drzew dla PT (10 realizacji)
tab.new <- sapply(vals, function(v) replicate(10, bagging.own.pred(bagging.own(data, v), newdata)$err))
# Wyznaczanie wartości średnich i odchylenia dla PU
tab.m <- apply(tab, 2, mean)
tab.s <- apply(tab, 2, sd)
# Wyznaczanie wartości średnich i odchylenia dla PT
tab.new.m <- apply(tab.new, 2, mean)
tab.new.s <- apply(tab.new, 2, sd)
# Tworzenie wykresów
errbar(vals, tab.m, tab.m + tab.s, tab.m - tab.s, ylim = c(0.3, 0.55))
errbar(vals, tab.new.m, tab.new.m + tab.new.s, tab.new.m - tab.new.s, add = T, col = "red", errbar.col = "red")
abline(h = err0, lty = 2)
abline(h = err1)
abline(h = err2, col = "red")
Rysunek 7.3
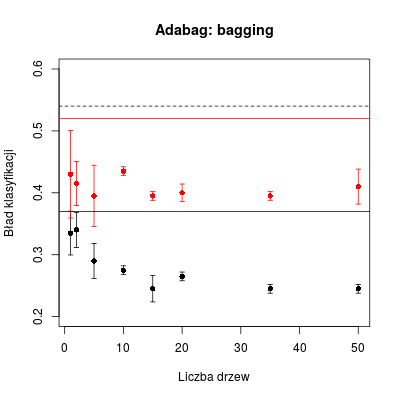
Jedną z dostępnych bibliotek implementującą algorytm baggingu jest adabag. W jej ramach działają funkcje bagging() oraz predict.bagging(), za pomocą których można wykonać obliczenia podobne do tych powyżej. Ze względu na trochę wolniejsze działanie dokonamy symbolicznych uśrednień (po 2).
library(adabag)
# Algorytm bagging
do.bag <- function(data, newdata, n) {
bag <- bagging(class ~ ., data = data, mfinal = n)
bag.pred <- predict.bagging(bag, newdata = newdata)
return(bag.pred$error)
}
# 2 realizacje bagging dla PU i PT
tab.bag <- sapply(vals, function(i) replicate(2, do.bag(data, data, i)))
tab.bag.new <- sapply(vals, function(i) replicate(2, do.bag(data, newdata, i)))
# Wyznaczanie wartości średnich i odchylenia dla PU
tab.bag.m <- apply(tab.bag, 2, mean)
tab.bag.s <- apply(tab.bag, 2, sd)
# Wyznaczanie wartości średnich i odchylenia dla PT
tab.bag.new.m <- apply(tab.bag.new, 2, mean)
tab.bag.new.s <- apply(tab.bag.new, 2, sd)
# Tworzenie wykresów
errbar(vals, tab.bag.m, tab.bag.m + tab.bag.s, tab.bag.m - tab.bag.s, ylim = c(0.2, 0.6), xlab = "Liczba drzew", ylab = "Bład klasyfikacji")
errbar(vals, tab.bag.new.m, tab.bag.new.m + tab.bag.new.s, tab.bag.new.m - tab.bag.new.s, add = T, col = "red", errbar.col = "red")
abline(h = err0, lty = 2)
abline(h = err1)
abline(h = err2, col = "red")
Rysunek 7.4
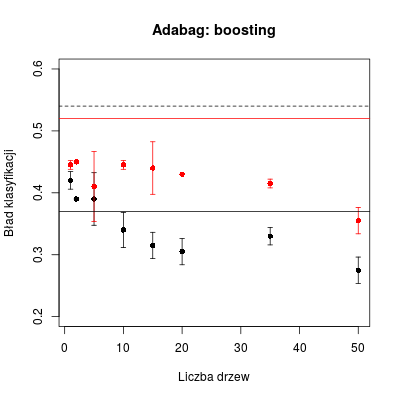
BOOSTING
Metoda boosting, choć w swojej konstrukcji podobna do baggingu, jest oparta na trochę innym pomyśle: kolejne klasyfikatory są uczone na zbiorze danych, w których prawdopodobieństwo wylosowania obserwacji jest zwiększane, jeśli w poprzednim kroku została ona źle zaklasyfikowana. Początkowo wszystkie wagi \(w_i\) $i=1,...,N$ są ustawione jako \(1/N\) (\(N\) - liczba elementów PU). W każdym kroku $m=1,..,M$ na danych trenowany jest przy użyciu wag \(w\) klasyfikator \(f_m\), a następnie zaś jest obliczana dla niego ważona ilość błędów
\(\mathrm{err}_m = \sum\limits_{i=1}^{i=N}w_i \mathrm{I}[y_i \ne f_m(\mathbf{x}_i)])\),
które są wykorzystywane do zmiany wag
\(w_i = w_i \exp(\gamma_m \mathbf{I}[y_i \ne f_m(\mathbf{x}_i)])\),
gdzie \(\gamma_m = \frac{1-\mathrm{err}_m}{\mathrm{err}_m}\); wagi są w międzyczasie normowane do jedności. Na końcu co do każdej obserwacji podejmowana jest decyzja zgodnie z regułą
\(\mathrm{sgn}\left[ \sum\limits_{m=1}^M \gamma_m f_m(\mathbf{x})\right]\).
W pakiecie adabag do boostingu są dedykowane funkcje boosting() oraz predict.boosting().
# Algorytm boosting
do.boost <- function(data, newdata, n) {
boost <- boosting(class ~ ., data = data, mfinal = n, control=rpart.control(maxdepth = 1))
boost.pred <- predict.boosting(boost, newdata = newdata)
return(boost.pred$error)
}
# 2 realizacje boosting dla PU i PT
tab.boost <- sapply(vals, function(i) replicate(2, do.boost(data, data, i)))
tab.boost.new <- sapply(vals, function(i) replicate(2, do.boost(data, newdata, i)))
# Wyznaczanie wartości średnich i odchylenia dla PU
tab.boost.m <- apply(tab.boost, 2, mean)
tab.boost.s <- apply(tab.boost, 2, sd)
# Wyznaczanie wartości średnich i odchylenia dla PT
tab.boost.new.m <- apply(tab.boost.new, 2, mean)
tab.boost.new.s <- apply(tab.boost.new, 2, sd)
# Tworzenie wykresów
errbar(vals, tab.boost.m, tab.boost.m + tab.boost.s, tab.boost.m - tab.boost.s, ylim = c(0.2, 0.6), xlab = "Liczba drzew", ylab = "Bład klasyfikacji")
errbar(vals, tab.boost.new.m, tab.boost.new.m + tab.boost.new.s, tab.boost.new.m - tab.boost.new.s, add = T, col = "red", errbar.col = "red")
abline(h = err0, lty = 2)
abline(h = err1)
abline(h = err2, col = "red")
Rysunek 7.5
LASY LOSOWE
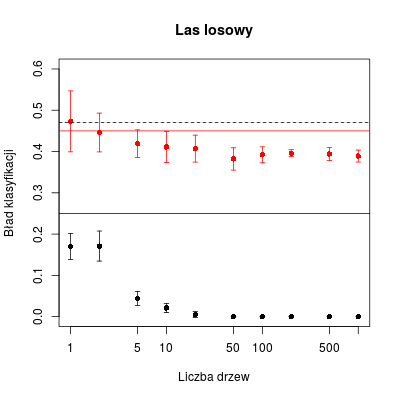
Ostatnim opisywanym algorytmem są lasy losowe. Tu także za każdym razem dokonujemy bootstrapingu, losując elemnty z PU do pseudopróby. Różnica polega na tym, że w każdym węźle drzewa wybiermay jedynie \(r\) spośród \(p\) składowych, na których dokonywany jest podział, przy czym sugeruje się \(r = \sqrt{p}\). Na koniec znów dokonujemy ostatecznej klasyfikacji zgodnie z większością głosów.
Wykorzystamy pakiet randomForest wraz z jego funkcją randomForest().
library(randomForest)
# Algorytm lasu losowego
do.rf <- function(data, newdata, n) {
rf <- randomForest(class ~ ., data = data, ntree = n)
rf.pred <- predict(rf, newdata = newdata)
return(err.rate(newdata$class, rf.pred))
}
# Parametry dla 10-wymiarowego rozkładu Gaussa
n1 <- 50
n2 <- 50
m1 <- rep(-1, 10)
m2 <- rep(1, 10)
S1 <- matrix(0, 10, 10)
diag(S1) <- 10
S2 <- S1
# Ustawienie ziarna dla losowania danych
set.seed(1290)
data <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
newdata <- draw.data.gauss(S1, S2, m1, m2, n1, n2)
colnames(data) <- c("X1", "X2", "X3", "X4", "X5", "X6", "X7", "X8", "X9", "X10", "class")
colnames(newdata) <- c("X1", "X2", "X3", "X4", "X5", "X6", "X7", "X8", "X9", "X10", "class")
vals <- c(1, 2, 5, 10, 20, 50, 100, 200, 500, 1000)
# 10 realizacji lasu losowego dla PU i PT
tab.rf <- sapply(vals, function(i) replicate(10, do.rf(data, data, i)))
tab.rf.new <- sapply(vals, function(i) replicate(10, do.rf(data, newdata, i)))
# Wyznaczanie wartości średnich i odchylenia dla PU
tab.rf.m <- apply(tab.rf, 2, mean)
tab.rf.s <- apply(tab.rf, 2, sd)
# Wyznaczanie wartości średnich i odchylenia dla PT
tab.rf.new.m <- apply(tab.rf.new, 2, mean)
tab.rf.new.s <- apply(tab.rf.new, 2, sd)
# Tworzenie pełnego drzewa
tree0 <- rpart(class ~ ., data, minsplit = 0, minbucket = 0, cp = 0)
# Przycinanie drzewa
tree <- prune(tree0, cp = choose.cp(tree0))
err0 <- err.rate(newdata$class, predict(tree0, newdata, type = "class"))
err1 <- err.rate(data$class, predict(tree, data, type = "class"))
err2 <- err.rate(newdata$class, predict(tree, newdata, type = "class"))
# Tworzenie wykresów
errbar(vals, tab.rf.m, tab.rf.m + tab.rf.s, tab.rf.m - tab.rf.s, ylim = c(0, 0.6), xlab = "Liczba drzew", ylab = "Bład klasyfikacji", log="x")
errbar(vals, tab.rf.new.m, tab.rf.new.m + tab.rf.new.s, tab.rf.new.m - tab.rf.new.s, add = T, col = "red", errbar.col = "red")
abline(h = err0, lty = 2)
abline(h = err1)
abline(h = err2, col = "red")
title("Las losowy")
Rysunek 7.6